DeepInsight: A methodology to transform a non-image data to an image for convolution neural network architecture - Scientific Re
It is critical, but difficult, to catch the small variation in genomic or other kinds of data that differentiates phenotypes or categories. A plethora of data is available, but the information from its genes or elements is spread over arbitrarily, making i
www.nature.com
이 논문에서 나온 접근법으로, 논문상에서는 gene expression 을 사용하여 2D image 로 만들어서 classification 에 사용함.
이런 접근이 없는 것은 아니지만 많이 있진 않은데, 내가 이전에 내 연구에 적용해봤을 때는 그렇게 좋은 성능을 나타내지는 못하였다. 하지만 아미노산의 one hot encoding 이나 dictionary 로 쓴다면 좋은 성능을 낼 수 있지 않을까.
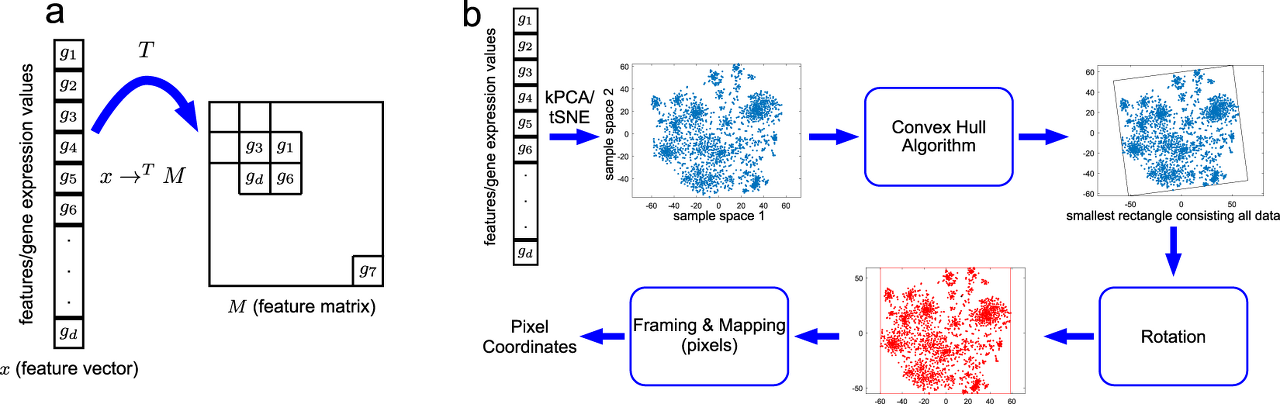
위의 그림에 나온 방법인데, 이 방법을 수정해서 사용하면 될 것 같다.
** 이 논문은 matlab 으로 구현되어 있어서, python 으로 다시 써야하나 검색해보았는데,
역시 구글신은 위대하다. Kaggle 에서 어떤 분이 해 놓으신 코드가 있다.
DeepInsight: Transforming Non-image data to Images | Kaggle
DeepInsight: Transforming Non-image data to Images
Explore and run machine learning code with Kaggle Notebooks | Using data from multiple data sources
www.kaggle.com
'Resources > Personal Projects' 카테고리의 다른 글
| [UniProt Challenge] Amino acid binding / pocket signature (0) | 2022.09.15 |
|---|---|
| [UniProt Challenge] PSSM : Binding residue representation (1) | 2022.09.15 |
| [UniProt Challenge] Key words (0) | 2022.07.10 |
| [UniProt Challenge] Reference (0) | 2022.07.06 |
| [UniProt Challenge] 1. Kick-off meeting (0) | 2022.07.04 |


댓글