Noninvasive Lung Cancer Early Detection via Deep Methylation Representation Learning | Proceedings of the AAAI Conference on Artificial Intelligence
2022, AAAI
AnchorDx Medical
기존의 Methylation 을 이용한 cancer detection 방법은 tissue level 의 방법들이 많아서, cfDNA 에 적용하였을 때 discrepancy 가 많았음.
따라서 이 논문에서는 cfDNA ( blood ) -based 방법을 만들고자 함

Dataset : 450K ( Illumina ) microarray
Number of samples : 424 ( Train : Validation : Test = 7:1:2 )
Model : Autoendoder ( methylation representation per region, per sample ) + classifier ( 여러가지 모델 사용 )
모델링에서 가장 중점이 되었던 개념은,
< Methylation 의 region 을 개별적으로 학습한 후 그것을 이어붙혀서 각 샘플의 representation 으로 만든다> 는 것.

위의 그림과 같은데, Ri 는 i번째 region 을 의미하고 그 안에 들어있는 여러가지 선은 read 를 의미한다.
450K 는 microarray 기반의 데이터이고, 이는 각각의 빛 신호를 read 로 말한 것 같다.
각각 methylation region에 들어있는 signal 을 autoencoder 에 학습시킨 후, i개의 z 라는 representation 을 만들어서 각 샘플(424개) 를 대표하는 matrix 를 만들어서 그 후 CNN, MLP ... 등의 classifier 에 feed 한 셈.

Autoencoder의 구조는 다음과 같은데, ResNet 의 구조를 차용했다고 한다.
Loss 함수는 일반적인 MSE 함수를 사용했다고 나오고, 이를 구하기 위해서 Convolution filter, reverse convolution filer 두 개를 사용해서 representation 을 만들기 전, 후 를 비교하였다.
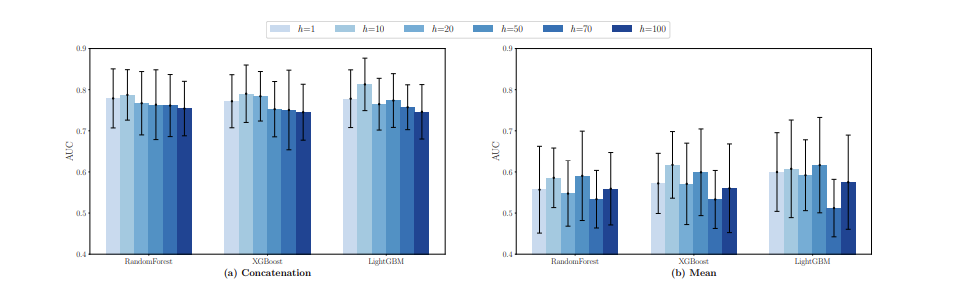
autoencoder 에서 나온 z 라는 각 methylation site의 representation 이 있고, 이 값을 이어붙혀서 논문에서 주요 논지 중 하나인 'Vectorize' 시킨 것과, 통상적으로 많이 쓰는 'Scalar' 로 사용하였을 때 Lung cancer detection 의 결과를 보여주는 결과로, 여기서는 RF, XGB, LightGBM 을 사용하였다.
Concatenate ( Vectorize ) 시켰을 때 성능이 훨씬 좋은 것을 볼 수 있고,
이 중에서도 region vector 를 10 개 사용하였을 때 제일 좋은 성능을 내는 것을 볼 수 있다.
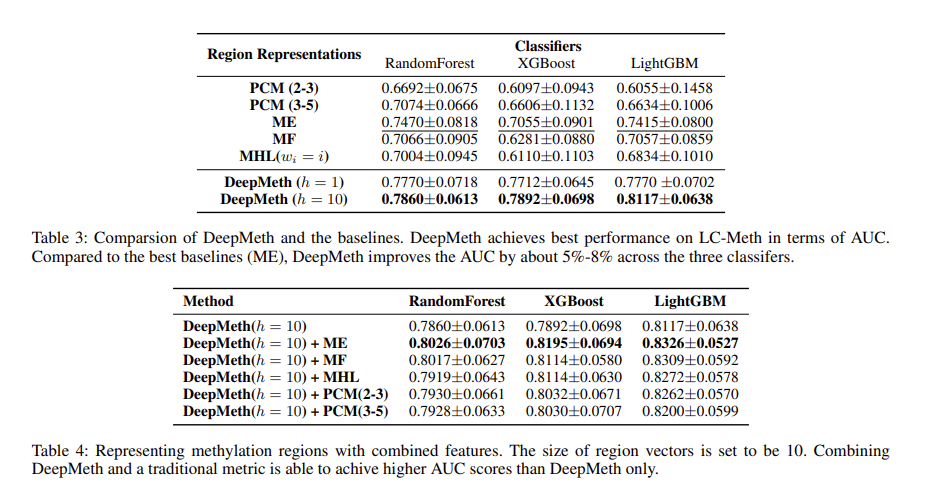
위의 표는 많이 쓰는 methylation 지표와 현 논문의 성능을 비교한 것이고 ( Table 3 )
아래 표는 그 두개를 합쳐서 결과가 얼마나 좋아지는가를 비교한 것이다 ( Table 4 ) .
두 경우 모두에서 최대 5-8% 정도의 성능 향상을 보였다.
2022년에 AAAI 에 나온 논문이고 Illumina 450K methylation microarray 데이터를 이용한 논문.
GRAIL 등의 회사에서 cfDNA WGMS 를 이용해서 조기진단제품을 만들어가고 있는 시대에서 450K는 타겟이 적어보일수도 있지만 기존의 beta value의 대체로 microarray signal 을 vectorize 해서 사용한다는 점이 좋은 접근인 것 같다.
모델링에 있어서 데이터가 굉장히 중요한데, protein sequence 나, 이러한 scalar value 인 경우에 특히 그런 것 같다.
microarray 데이터를 사용함에 있어서 noise reduction tech 도 들어갔을 것 같은데, 이 논문에 언급하지는 않았고, 만약 사용하게 된다면 그 부분을 보완하여 사용하여도 될 것 같다.
Tissue 와 body liquid 에서의 biomarker 는 겹치지 않은 경우가 많은 것 같다. 기존의 pre-define 된 지역을 보는 것 보다는 조금 더 넓은 영역 ( methylation site 등 ) 을 보면 searching space 가 늘어남에 따라 새로운 마커나, unified dx model 를 찾을 수 있지 않을까 기대해 본다.


댓글