2023, Cell Reports Medicine
Michael Hoffmeister, Jakob Nikolas Kather Groups

Abstract
- 딥러닝은 CRC 에서 histopathology 를 가지고 MSI 를 예측할 수 있지만, 다른 바이오마커도 예측할 수 있을지는 미지수임.
- 이 논문에서 2개 큰 코호트에 대해서, 6개의 최신 딥러닝 구조들을 사용하여 MSI, BRAF, KRAS, PIK3CA 같은 mutation 예측의 성능을 비교함.
- Self-supervised, attention-based multiple instance learning 이 성능이 가장 좋았고, 설명가능성도 제공했음.
- MSI 하고 BRAF 의 예측은 임상에서 사용가능한 결과를 냈지만 PIK3CA, KRAS, NRAS 의 예측의 경우 충분하지 않았음.
M&M
Target value processing

Image processing
All images from H&E stained resection tissue slides were preprocessed according to the ‘‘Aachen protocol for deep learning histopathology’’.
- Tesselation : WSI 는 512*512 픽셀로 잘라짐. ( 256mm edge length )
- Tissue selection : RGB thresholding 을 이용해서 자동으로 선택 + image tile 당 적어도 4개의 edge 를 요구하는 Canny edge detection 을 사용하여 선택. 모든 tile 들 사용
- Tile processing : Bilinear interpolation 이 구현되어 있는 PyTorch 의 Resize() 를 이용하여 224 px edge length 로 처리된 후 ImageNet의 RGB 픽셀 값의 평균, 표준편차로 정규화됨
Bilinear interpolation : 이미지를 continuous 하고 smooth 하게 하기 위한 resampling 방법.
Why 224 pixel? : 224 pixel image 는 딥러닝에서 standard size 로 선택된 사이즈로, image resolution, computional efficiency 를 고려한 사이즈임. 이는 딥러닝 모델의 input 이 일정하게 하고 다른 이미지들과 비교를 수월하게 함.
Resample 을 하는 이유? : Resample 의 결과 이미지의 크기가 바뀌거나, 해상도가 바뀜. 이는 이미지를 어떤 기준이나 요구사항에 맞추기 위함임. 딥러닝 모델의 경우, resampling 은 input size 를 표준화시키기 위해서 많이 사용됨.
- Color Normalization : Training set 의 타일들은 reference image tile 을 사용하는 Macenko 의 방법으로 color-normalize 됨. Test set 에서는 Native tile, color-normalized tile 별로 성능을 따로 구함.
Biomarker prediction from whole slide images
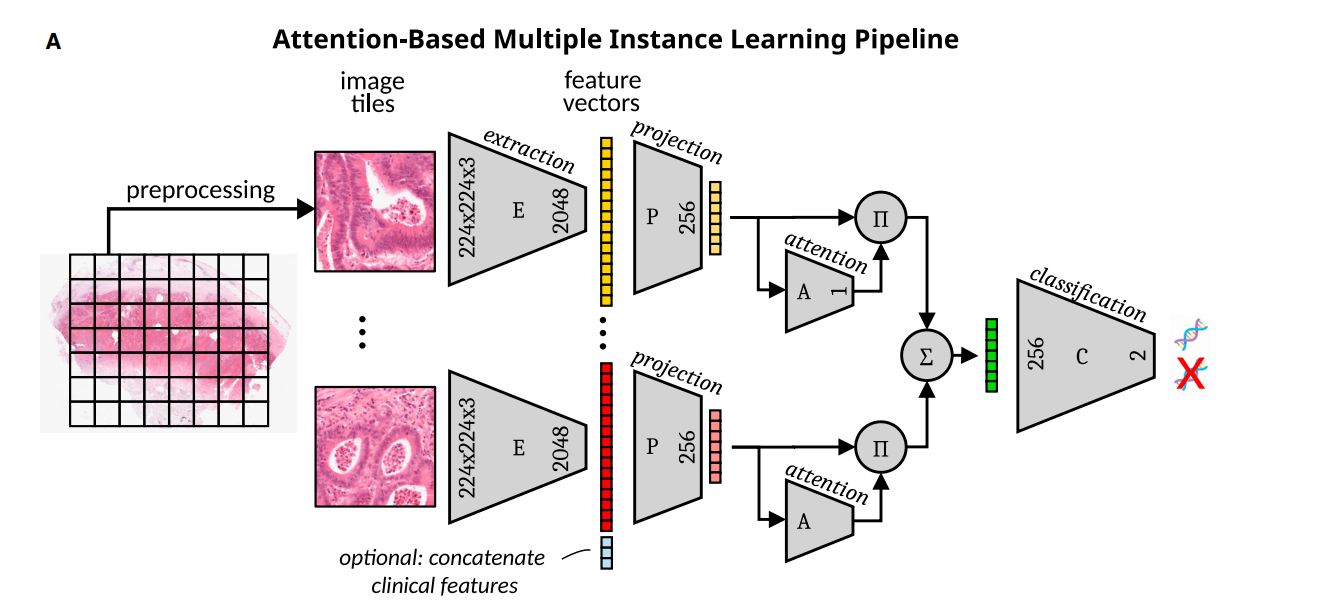
구조를 말해주고 있음
attMIL, Attention-Based Multiple Instance Learning 은 여러 instance ( 여기서는 여러개의 tile 을 말함 ) 을 가지고 예측하는 방법을 말하는 것으로, 보통 instance 는 딥러닝에서 하나의 input 을 말함.
- 두개의 DL 접근법을 비교함. ImageNet pretrain ( INPT ) approach, Attention-based Multiple Instance Learning ( attMIL ) 이 그것임.
- INPT 에서는 slide 수준의 예측을 모든 타일 예측치의 평균으로 진행했는데, 외부 코호트에서 완벽하게 일반화되지는 않았음.
- attMIL 방법은 두 단계로 이루어졌음 : 먼저, 타일들은 pretrained encoder 를 이용해서 image feature vector 로 압축됨. 그 후 image feature vector 는 attention mechanism 을 이용하는 network 의 input 으로 들어가고 slide level 의 prediction 을 도출 ( 어떤 tile 이 최종 prediction 에서 제외되어야 하는지를 학습 )
- 모델을 공개되어있는 2 개의 frozen (pretrained) Encoder 로 학습하였는데 ( Ciga et al., Wang et al., ) 이 둘은 모두 Self-supervise learning 임. ( 이를 SSL-attMIL ) 이라고 함
- Ciga et al. applied SimCLR60 to train a ResNet-18 on 400,000 pathology images selected from 57 datasets.
- Wang et al. trained a ResNet-50 on a total of 15 million pathology images retrieved from 32,000 WSIs from the full TCGA and PAIP dataset via a clustering-guided contrastive learning (CCL) SSL algorithm.
- CCL 에서는 같은 WSI 에서 두 개의 다른 tile 간에 contrastive loss 를 최소화하고, 다른 WSI 와 비교해서 tile 간의 loss 를 최대화하는 것.
- SimCLR 에서는, 같은 tile 에 대해서 contrastive loss 가 최소화되고, 다른 tile 간에는 최대화 됨.
- CCL, SimCLR 각 tile 별로 1024, 2048 의 feature 를 뽑았고 한 WSI 당 랜덤하게 512 개의 feature 를 선택하여 attMIL model input 으로
- attMIL 모델 + clinical data 해서 확장함 ( 주로 OHE ), 각 tile 에 붙이는 식으로.
Implementation of the INPT approach
- 먼저 tranfer learning 으로 학습했는데, 이는 ImageNet 으로 학습된 CNN 모델을 필요로 했음. 따라서 먼저 new classification head 의 weight 빼고 다 고정시켜 놓은 후, 그 다음에 나머지 layer 의 weight 들이 fine-tune 되었음.
- 환자 수준의 prediction 은 tile 의 prediction value 를 평균하는 것으로.
- in-house open-source pipeline DeepMed , batch size of 92, the Adam optimizer (b1 = 0:9;b2 = 0:99;ε = 10^- 5), and a learning rate of 2e-3 and 1% weight decay.
- The cross-entropy loss function was weighted by the inverse of class frequencies to account for class imbalances.
- 모델 classification head 의 fine tuning 이 하나의 에폭에서 끝났을 때, 전체 model 은 32 에폭으로 train 됨. 과정동안 Learning rate 은 증가되었다가 감소되는 형식으로 사용되는데, 이를 One-cycle policy 라고 함.
- beta1 도 첫 번째 10 epoch 동안은 0.95~0.85 로 바뀌고 나머지 epoch 에서는 0.95 로 다시 돌아옴.
- Training 시 tile 은 random rotation 을 통해서 augmented 됨.
Implementation of attention-based multiple instance learning
- 두 개의 SSL-attMIL 방법 모두에서, 타일 k 에 대해서 ReLU 뒤에 오는 FC layer는 256차원으로 feature 를 embed 하고, 이 embed 된 vector 는 다른 256개 vector ( hk ) 를 만드는 선형 layer 로 들어감.
- K 번째 tile 에 대한 attention score ak 는 이렇게 구해짐.


- MIL pooling 수식은 다음과 같음 :

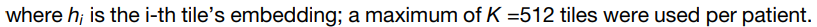
- 이렇게 계산된 hsum ( 하나의 WSI 에서 attention 을 거쳐서 나온 하나의 값 ) 들은 batch 로 BatchNorm1D layer, followed by Dropout layer with p=50% 를 거친 후 WSI 당 2개의 output 을 뽑아내는 FC layer 로 전달되고, 맨 마지막에 softmax 를 통해 최종 결과를 얻게 됨.
- The batch size was 32 patients, the number of epochs was 32
Result

성능을 나타내고 있고, clinical 정보가 합쳐진 결과 ( one hot encoding 등을 통해 tile-derived feature 에 concat 된 input ) 을 이용했을 때 결과가 좋게 나오는 것도 확인가능함.

Normalized attention ( minmax ) , predicted BRAF score 를 비교한 결과.
Attention 이 강한 지역에서는 BRAF score 도 강했다는 이야기
Kather Lab 에서는 주로 attMIL 을 이용해서 여러가지 다른 형질을 예측하는 논문을 냈고,
MSS 예측은 19' Nature medicine 에 나온 내용.
이번 논문에서는 대표적인 oncogenic mutation 을 예측하려 했지만 BRAF 이외에서는 힘들었다고 이야기하고 있고, mutation 의 종류가 나와있지 않은 것으로 보아서 BRAF, KRAS, PI3KA 등의 hotspot mutation 을 포함한 모든 mutation 이 포함되는 듯 하다.
- 환자가 너무 많긴 했지만 특정 위치의 mutation 만 target 으로 했으면 더 성능이 잘 나왔을지도?
- 흔히 hotspot mutation 이라 함은, 발암 유전자에서 driver mutation 이라고도 볼 수 있으므로, 이들이 주는 impact 가 더 커서 더 좋은 target 이 되지 않을지?
Reference
Attention-based Deep Multiple Instance Learning (arxiv.org)
Xiyue-Wang/RetCCL (github.com)
KatherLab/marugoto: Tools to build deep learning pipelines. (github.com)
KatherLab/deepmed (github.com)
marrlab/attMIL: Attention Based Multiple Instance Learning (attMIL) (github.com)


댓글