Nature Genetics, 2023
Pablo Porras & Pedro Beltrao Labs
Abstract
상호작용하는 단백질은 비슷한 기능을 가지는 경향이 있으며, 이는 같은 기관적인 형질에도 영향을 미친다.
상호작용 네트워크는 GWAS 연구로부터 후보 형질-연관 유전자를 확장하는 데 사용될 수 있다.
이 연구에서는 1002개의 인간 형질에 대한 형질-연관 유전자의 네트워크 기반 확장을 통해 이것이 알려진 질병 유전자와 약물 타겟 유전자로까지 회복(recover) 될 수 있음을 보였다 (역주 : GWAS 에서 관계되어있지 않은, 혹은 놓친 gene 을 다시 recover함).
네트워크 확장 점수의 유사성은 생물학적/유전적 특징을 공유하고 있는 형질의 그룹을 식별한다. 이 연구에서 여러가지 형질에 연관되어있는 73개의 다면 발현 유전자의 module 을 식별하였고, 이들은 protein ub, RNA processing 같은 process 와 연관되어 있었다.
유전자 제거 연구들과 반대로, 여기서 정의되는 pleiotropy는 여러가지 세포가 연관되는 process 를 말한다. 우리는 이 연구에서 알려진 pathogenic variant 를 가지고 있어 약물 타겟으로 사용될 수 있는 유전자가 enrich 된 인간 질병 연관 module 의 예시를 보였다.
마지막으로, 우리는 염증성 장 질환 GWAS loci에서 유전자를 연구하기 위해 네트워크 확장 점수의 사용을 설명하고, 강력한 기능 및 유전적 인 증거을 가진 염증성 장 질환 관련 유전자를 제시한다.

네트워크 구성과 gene mapping
1. 네트워크의 구성을 위해 PPI(Protein-protein interaction) database 를 다 합쳐서 사용했음. (FigA)
2. 또한 Open Targets Genetrics 의 locus-to-gene(L2G) score 를 이용하여 GWAS 형질 연관성이 개별 gene 에 mapping 됨. 이 score 는 SNP fine-mapping, gene 거리, QTL 정보등을 사용하여 원인유전자를 찾는 기계학습론적 방법임.
3. 각각의 GWAS 에 대해서, 연관유전자들은 network 에서 seed 유전자로 사용됨. 적어도 한 형질과 연관되어있는 7660 개 GWAS 유전자 중 7248 개가 network에 있는 단백질과 상응했음.
4. 그 후 Personalized PageRank(PPR) 를 사용하여 네트워크 안에 있는 모든 protein-coding 유전자들에 대한 점수를 구함. 이것을 network propagation score 라고 함. 이 점수는 GWAS 연관 유전자와 연결되어 있는 경로가 짧을수록 ( 가까이 있을수록 ) 점수가 높이 매겨짐.
5. network propagation score 상위 25% 에 있는 유전자들을 가지고 gene module 식별함. 이 유전자들 중에서 high network propagation score(Kolmogorov–Smirnov test 를 이용한 BH-adjusted P<0.05) 과 GWAS-연관이 두 경우 이상인 경우임.
6. 이 접근법을 1002개 형질에 적용했는데, 이는 interactome 에 두 개 이상 gene 이 mapping 된 경우임. GWAS 데이터는 21개 therapeutic area를 cover 하며 GWAS-연관 유전자의 수는 2-763 개였음(median 6개)
만들어진 network 검증
형질 연관 유전자를 recover 하는 network 확장 기법의 효용성을 알아보기 위해, 우리는 인간 질병의 약물 타겟이 되는 유전자 set 을 질병 관련 유전자들의 golden standard 로 정의했음.
벤치마킹 시 circularity 를 피하기 위해. 각 질병에 대하여 GWAS-연관 유전자 중 golden standard 유전자와 겹치는 유전자들은 제외시켰음.
Network propagation score 를 이용하여 예측을 진행한 결과 AUC>0.7 이상의 성능으로 질병 연관 유전자를 예측함. 이는 golden standard 유전자 set 을 이용한 random permutation 보다 좋은 결과임. 이는 golden standard gene 에 의해서 크게 biased 되지 않는다고 말할 수 있었음. 다른 PPI net 을 이용해본 결과 결과가 더 좋거나 비슷했음.
결과적으로 1002 형질에 대한 network propagation score 와 906 형질에 대한 gene module 을 구했음.
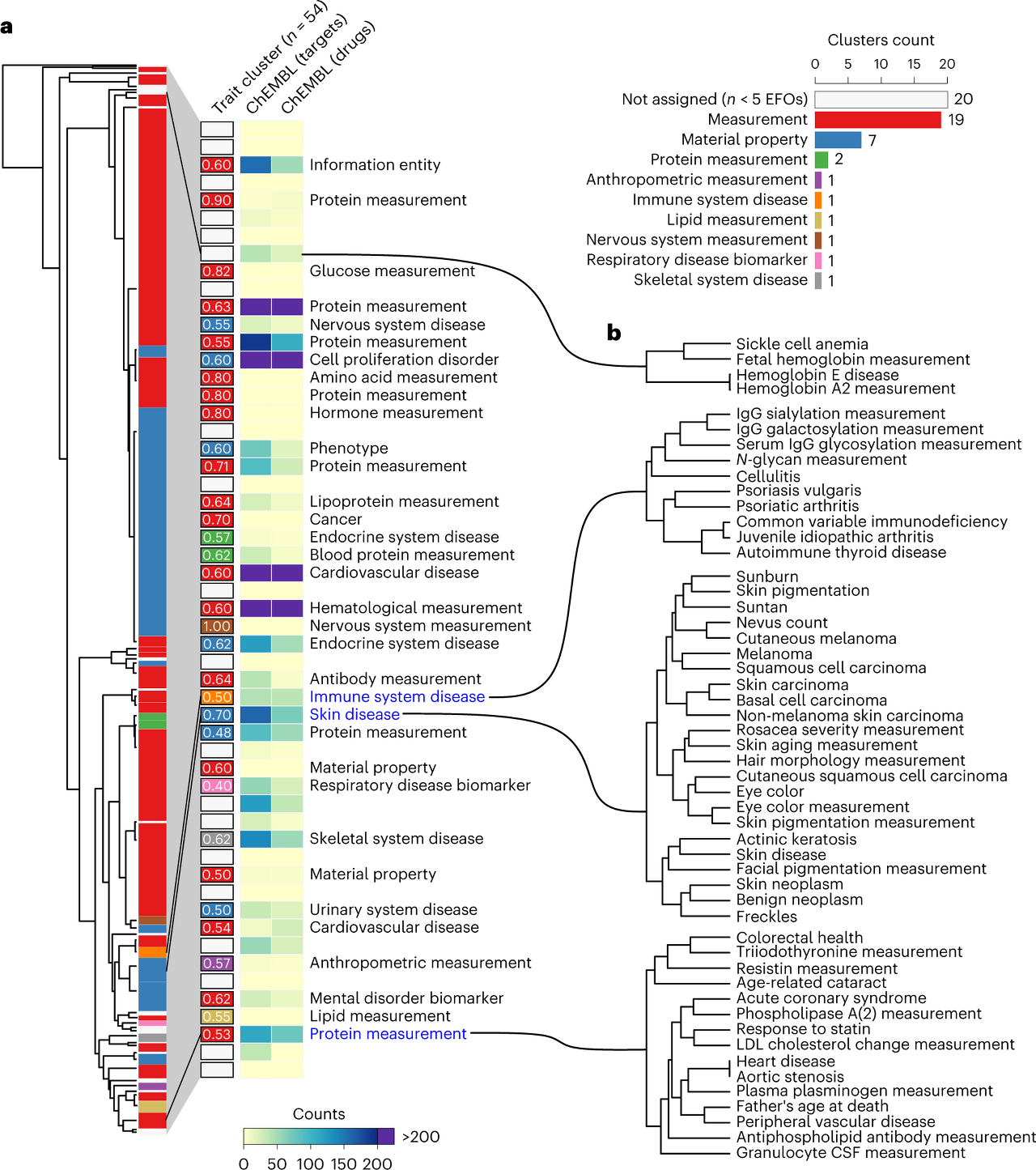
연관 형질 그룹의 식별
하나의 질병을 치료하기 위해 사용되는 약물이 관련 질병에도 영향을 미칠 수 있기 때문에 공통의 유전적 기반을 가질 가능성이 있는 형질 그룹을 식별하는 것은 가치가 있음.
인간 형질의 유전적인 공유는 GWAS 에서 유도되는 SNP 수준의 연관성에서 도출되는 경우가 많으나 이 접근법은 공유 유전학이 공유된 생물학적 과정에 어떻게 대응하는지를 식별하지 못함.
또한 많은 GWAS는 이러한 비교에 필요한 전체 요약 통계를 보고하지 않음. 대조적으로, Network propagation 점수는 모든 GWAS에 사용 가능한 후보 유전자 세트에서 계산할 수 있음.
Network propagation에서 파생된 특성-특성 연관성을 벤치마킹하기 위해 질병 유형, 해부학 및 세포 유형의 측면을 포함하는 Experimental Factor Ontology(EFO) 의 주석 유사성을 사용함.
예를 들어, 비슷한 신경학적 특성들은 EFO에서 많은 주석 용어를 공유하는 경향이 있음.
이러한 주석을 사용하여 기능적으로 관련되어 있어서 공통의 유전적 기반을 가질 가능성이 있는 796쌍의 특성을 정의함.
교란된 생물학적 프로세스의 유사성을 기반으로 특성-특성 관계를 탐색하기 위해 네트워크 전파 점수의 쌍별 거리를 사용하여 계층적 클러스터링을 통해 트리를 구축하고(그림 2a) 54개의 특성 하위 그룹을 정의함
또한 ChEMBL 에서 각 cluster 의 질병에 대해서 drug indication 을 가져왔고, 이는 cluster 안에서 어떤 drug 들이 repurposing 될 수 있는지, 또 어떤 drug development 가 가장 필요한지를 찾을 수 있게 했음. 64개의 형질을 나타내는 18개의 클러스터는 drug 가 없었고, drug development 상에서도 많이 발달되어있지 않았음.
Pleiotropic Modules

인간 세포생물학에서의 pleiotropy 는 다른 많은 인간 형질과 관련되어있는 gene module 을 찾음으로서 연구할 수 있음. 이는 특정한 부분의 perturbation 이 어떻게 다양한 형질에 대해서 연관을 가지고 있는지에대한 이해를 도움.
총 2,021개의 유전자 모듈과 형질 간의 연관성을 발견하였으며, 이 중 886개(43.8%)는 단일 형질과 연관된 유전자 모듈이며, 나머지는 둘 이상의 형질과 연관된 73개의 유전자 모듈로 볼 수 있음.
하나 이상의 형질과 관련된 73개의 모듈은 유의하게 더 많은 유전자를 가지고 있지 않은 반면(P = 0.72, 콜모고로프-스미르노프 테스트), 형질 중73개의 다방성 유전자 모듈과 관련된 것들 은 유의한 초기 GWAS seed 유전자의 수가 더 많은 경향이 있었음.
따라서 많은 gene 과 관련되어있는 형질은 pleiotropic gene module 과 관련되어있을 확률이 높았음.
Pleiotropic gene module
연구에서 가장 pleiotropic 했던 모듈은 6개로, 56-110 개의 형질과 관련이 있었음.
Protein Ub, GPCR, RNA processing, ECM organization 등와 관련이 있었음.
효모에서의 유전자 제거 실험은 매우 pleiotropic 한 cellular process 를 밝혔음. 10개 이상의 형질과 연관된 pleiotropic module 의 유전자들은 보편적으로 발현되는 유전자들이었으며, 많은 종류의 deletion phenotype 을 가지고 있으며 genetic interaction 의 수가 많았음.
이러한 분석은 이 연구에서도 적용할 수 있었는데, 예를 들어, '자극 스펙트럼 장애' 및 '골관절염'과 같은 형질과 관련된 전능성 모듈은 단백질 수송에 영향을 미치는 유전자 삭제 표현형의 비율이 높고, 알츠하이머병, 대머리 측정 및 골밀도와 관련된 모듈은 세포 죽음와 관련된 유전자 삭제 표현형의 비율이 높은 유전자를 가짐.
그런 다음 여기서 파생된 모듈-특성 연관성에 의해 정의된 pleiotropy 을 CRISPR 유전자 삭제 연구에 의해 정의된 pleiotropy와 관련시킴.
각 유전자 온톨로지(GO) 용어에 대해, 우리는 분석에서 많은 특성과 연결된 유전자의 농축과 많은 유전자 삭제 표현형을 가진 유전자의 enrichment를 계산.
우리의 정의에 기초하여 다세포 유전자에서 특별히 풍부한 GO 용어는 막 신호 전달, 세포 간 통신 및 세포 이동과 같은 다세포성과 관련된 용어들.
CRISPR 스크린에서 특히 발견되는 전능성의 경우, 우리는 세포 주기, 리보솜 생합성 및 RNA 대사와 같은 필수 과정과 관련된 용어들.
공유된 Mechanism 을 이용한 drug repurposing
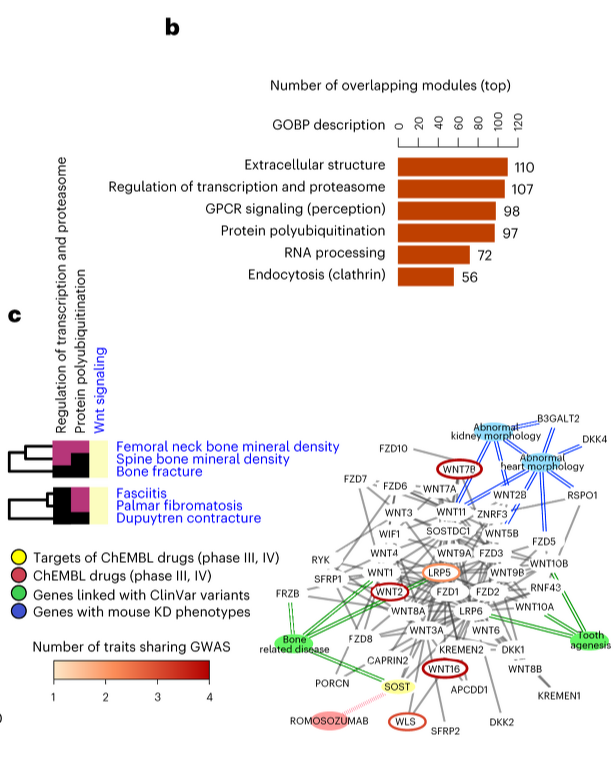
- 연구에서 두 개의 형질 ( bone, fasciitis )이 공통적인 gene module을 가질 것이라고 예상되었음.
- Wnt signaling gene 이 enrich 된 모듈이었는데 이는 bone, fasciitis 에 관련되어있다고 알려져있었음.
- ClinVar 에서 likely pathogenic 하다고 알려진 gene 을 모았고 이 모듈은 tooth agenesis, bone-related disease 를 가진 환자들에서 나타나는 pathogenic variant 의 gene 이 enrich 되어 있었음.
- 또한 이 module 은 osteoporosis 에 효과적이라고 알려진 drug 인 Romosozumab 의 target 도 가지고 있었음.
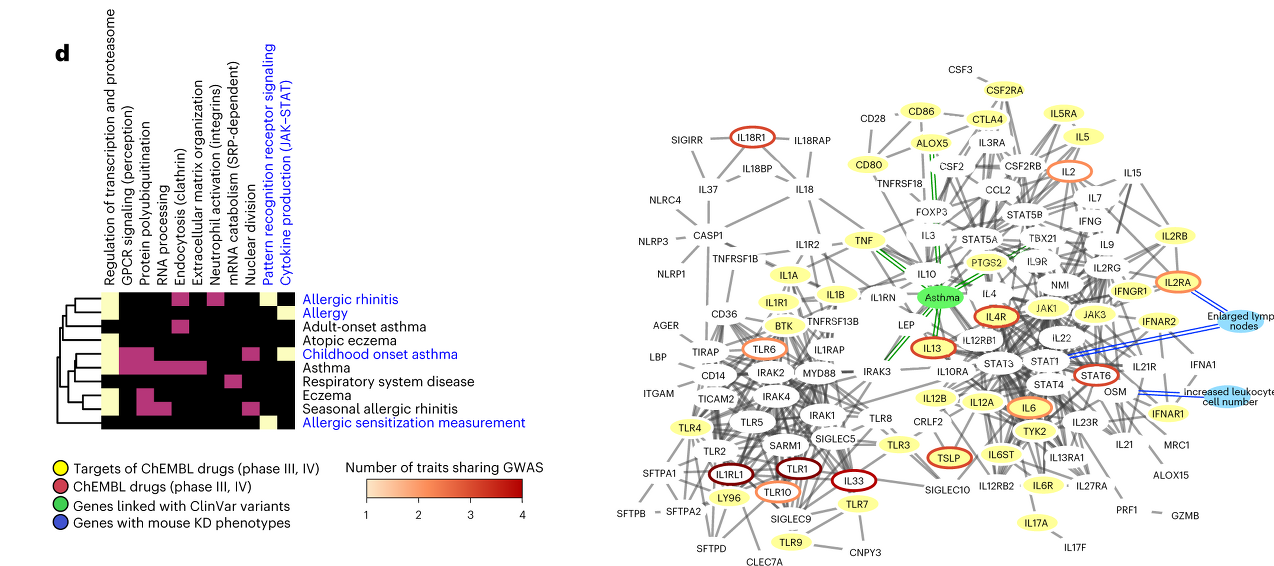
두 번째 예로 10개의 호흡기 질환과 면역질환이 연관된 그룹을 찾았음.이 그룹은 3개의 모듈로 구성.
regulation of transcription and proteasome, and two more specific modules related to pattern recognition receptor signaling and cytokine production with Janus kinase/signal transducer and activator of transcription (JAK–STAT) involvement.
이 3개의 모듈은 천식을 가진 환자로부터 온 pathogenic variant 의 gene들에 대해 굉장히 enrich 되어있었음.
위 그림에서 나타난 2개의 모듈에서는 GWAS 에서는 나오지 않았지만 pathogenic variant 를 가지는 gene 을 표시해둔 것 인데, 그 중 IRAK3 의 경우 GWAS 천식 연구에서는 나오지 않았지만 천식에 대한 mice 모델 연구에서 IRAK3 의 조절이 IL-33로부터 유도되었음을 말해주고 있음. 따라서 현재는 clinic 에서 쓰이는 약이 없지만, asthma 나 다른 호흡기 질환을 위한 target 이 될수도 있음을 시사함
면역관련 질환에 대한 gene 모듈들과 IBD
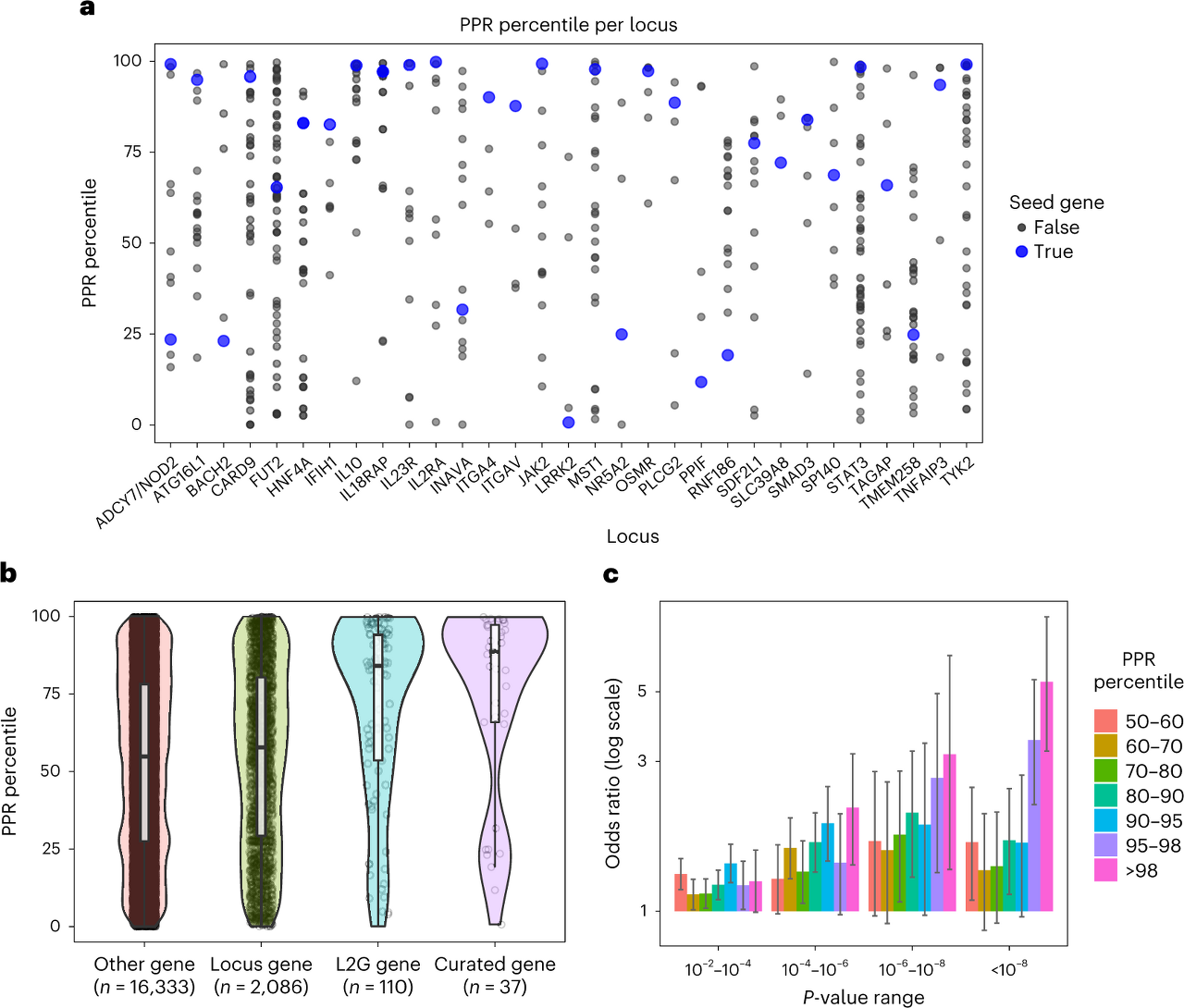
- 뭐니뭐니해도 seed gene 을 고르는 것이 가장 중요함.
- 여기서는 loci-level 로 고르는 예시를 보여줌.
- 따라서 GWAS-based 의 seed gene curation 과 L2G score 기반의 seed gene curation 을 비교한 결과 manual curation 이 결과가 더 좋았음. ( Network propagation score 가 더 높았음 )
- P value 는 약간 떨어지지만 L2G 기반의 점수도 높았음. ( c,d )
Discussion
내 요약
- 네트워크 확장은 GWAS loci에 없지만 동일한 생물학적 과정을 변조할 수 있는 유전자를 포함하여 GWAS에 의해 식별되지 않은 이전에 알려진 질병 유전자를 검색할 수 있다.
- 우리는 동일하거나 관련된 질병에 대한 임상 변이를 가진 유전자가 풍부한 질병 관련 유전자 모듈의 예를 보여주었다. 임상 변이를 가진 유전자가 GWAS 연계 유전자와 겹치지 않은 경우가 많았는데, 이는 임상 변이 빈도가 낮기 때문일 가능성
- 선택된 유전자 세트 내에서 기능 상실 변이체의 부담을 테스트하는 것은 low-frequency 변이체의 영향을 연구하는 데 사용되는 접근법이며, 우리는 여기서 식별된 유전자 모듈이 이러한 목적에 이상적일 수 있음을 제안
- 여기서 확인된 유전자 모듈은 서로 다른 인간 특성을 가진 세포 생물학의 특정 측면과 관련이 있다. 마우스 표현형과 ClinVar 변이형의 분석은 확인된 관계 중 일부에 대한 추가 증거를 제공했다. 특히 적절한 모델(예: 오르가노이드, 마우스 모델)을 사용하여 파생된 연관성의 일부를 추적하려면 추가 실험 작업이 필요
- 유전자 모듈을 식별하는 것 외에도, 우리의 GWAS 기반 네트워크 접근법은 IBD에 대해 보여준 것처럼 특정 생물학적 과정 내에서 그들의 역할에 따라 개별 위치에서 질병 유전자의 우선순위를 정하는 데 사용될 수 있다.
- 흥미롭게도, 높은 다방성 유전자 모듈과 관련된 형질은 보통 더 큰 샘플 크기를 갖는 시작 GWAS 씨앗 유전자의 수가 더 많은 경향이 있다. 이는 형질과 관련된 위치의 수가 많을수록, 그리고 샘플 크기가 클수록, 이 형질이 유전적으로 매우 다형성적인 생물학적 과정과 연관될 가능성이 높다는 것을 시사한다. 복잡한 형질의 유전성이 게놈 16을 따라 광범위하게 퍼져 있다는 것이 제안되었지만, 우리의 분석은 이 유전성이 많은 형질에 걸쳐 무작위적이지 않은 방식으로 겹친다는 것을 보여준다.
- 요약하면, GWAS의 네트워크 확장은 인간의 특성과 관련된 유전자 및 세포 과정을 식별하기 위한 강력한 도구이며, 다중 특성 분석의 적용은 유기체 수준에서 인간 생물학적 경로의 다원성을 드러낼 뿐만 아니라 약물 개발 및 용도 변경에 대한 새로운 기회를 강조할 수 있다.
ChatGPT 요약
본 연구는 GWAS의 네트워크 확장을 이용하여 906개의 인간 특성과 관련된 유전자 모듈을 확인하였다. 이 연구는 네트워크 확장이 GWAS loci에 없지만 동일한 생물학적 과정을 변조할 수 있는 유전자를 포함하여 GWAS에 의해 식별되지 않은 이전에 알려진 질병 유전자를 회수할 수 있다는 것을 발견했다. 식별된 유전자 모듈은 직접적인 유전자 지원이 있는 유전자를 제외하는 경우에도 성공적인 약물 표적을 위해 풍부하다. 그러나 이러한 유전자를 변조하는 영향의 방향에 대한 통찰력을 얻기 위해서는 추가 작업과 해석이 필요하다. 이 연구는 조직 또는 세포 유형별 네트워크를 도출하기 위한 매핑 적용 범위와 계산 또는 실험 접근 방식의 개선이 네트워크 확장의 미래 효과에 큰 영향을 미칠 수 있음을 시사한다.
이 연구는 동일하거나 관련된 질병에 대한 임상 변이를 가진 유전자가 풍부한 질병 관련 유전자 모듈의 예를 보여주었다. 서로 다른 특성과 연결된 유전자 모듈은 약물 용도 변경 또는 교차 질병 약물 개발을 위한 기회를 제공할 수 있지만, 다방성 프로세스를 대상으로 하는 것은 안전 문제를 제기할 수 있다. 연구 결과, 다성 유전자 모듈은 보편적으로 발현되는 유전자에 대해 풍부하고, 유전자 삭제 표현형이 많고 유전자 상호작용 횟수가 높은 것으로 나타났다. 흥미롭게도, 높은 다방성 유전자 모듈과 관련된 형질은 보통 더 큰 샘플 크기를 갖는 시작 GWAS 씨앗 유전자의 수가 더 많은 경향이 있다.
요약하면, 이 연구는 인간의 특성과 관련된 유전자와 세포 과정을 식별하기 위한 GWAS의 네트워크 확장의 힘을 강조하고, 약물 개발과 용도 변경을 위한 새로운 기회를 제안한다. 그러나, 그러한 유전자를 변조하는 것의 영향과 다방성 프로세스를 표적화하는 것과 관련된 안전 문제를 완전히 이해하기 위해서는 추가 작업이 필요하다.


댓글